
내가 보려고 만든 머신 러닝 기초 다잡기
데이터셋은 하단 URL을 통해 다운로드
https://www.kaggle.com/datasets/mathchi/diabetes-data-set
Diabetes Dataset
This dataset is originally from the N. Inst. of Diabetes & Diges. & Kidney Dis.
www.kaggle.com
작성 언어는 Python 3.8이며 주피터 혹은 캐글 노트북 등을 활용하거나, ipynb 확장자의 형태로 편집기를 활용해도 무방
그 외 pandas나 plot의 파라미터의 경우 하단 URL을 참고하였다.(매우 도움이 되었음!)
https://wikidocs.net/book/7188
# 0_데이터셋 살펴보기
해당 데이터셋은 Diabetes(당뇨병) 환자에 대한 데이터셋이다. 각 컬럼을 살펴보면 다음과 같다.(번역은 틀릴 수 있음..)
| Pregnancies | Number of times pregnant 임신한 횟수 |
| Glucose | Plasma glucose concentration a 2 hours in an oral glucose tolerance test 2시간 동안의 경구 포도당 내성 검사를 통한 혈장 포도당 농도? |
| BloodPressure | Diastolic blood pressure (mm Hg) 혈압 |
| SkinThickness | Triceps skin fold thickness (mm) 삼두근 피부주름 두께 |
| Insulin | 2-Hour serum insulin (mu U/ml) 2시간 동안의 인슐린 농도? |
| BMI | Body mass index (weight in kg/(height in m)^2) 우리가 흔히 아는 BMI |
| DiabetesPedigreeFunction | Diabetes pedigree function 당뇨병 가족력 함수? |
| Age | Age (years) 나이 |
| Outcome | Class variable (0 or 1) 당뇨병 여부 |
이제 코드로 해당 데이터를 살펴보겠다.
# 1_데이터 불러오기
코드 작성이 편리하도록 해당 데이터셋의 컬럼명을 변경한 뒤 시작하였다.
import numpy as np
import pandas as pd
filename = 'diabetes.csv'
data = pd.read_csv(filename)
data.columns = ['preg', 'plas', 'pres', 'skin', 'test', 'mass', 'pedi', 'age', 'class']
peek = data.head(20)
print(peek)
print(data.shape)
출력과 같이 총 768행 9열로 이루어져 있다.
# 1-1_데이터 타입 확인
data.dtypes
# 또는
#print(data.dtypes)
# 1-2_출력 데이터 표시 형식 변경 및 데이터 요약 출력
pd.set_option('display.width', 100) # 한 행에 출력할 컬럼 갯수(100개)
pd.set_option('display.precision', 3) # 소수점 셋 째자리 까지만 표기
data.describe() # 데이터 요약 출력
*(참고 / count: 데이터 개수, mean: 평균, std: 표준편차, min: 최소값, 25 ~ 75%: 백분위 값, max: 최대값)
# 1-3_컬럼의 데이터값 별 분포 확인
class_counts = data.groupby('class').size()
print(class_counts)
데이터의 라벨 등을 확인할 때 유용할 듯?
데이터 상의 당뇨병 환자는 전체에서 268명
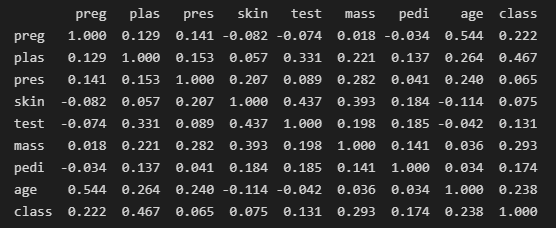
# 2_데이터 상관관계 분석
correlations = data.corr(method = 'pearson')
print(correlations)method는 총 세 가지(pearson / kendall / spearman)
| pearson 피어슨 상관계수 |
두 변수 간의 선형 상관관계를 계량화 한 수치. 코시-슈바르츠 부등식에 의해 +1과 -1사이의 값을 가짐. +1의 경우 완벽한 양의 선형 상관 관계, -1의 경우 완벽한 음의 상관관계, 0의 경우 선형 상관관계를 갖지 않음 |
| kendall 켄달-타우상관계수 |
두 변수들간의 순위를 비교해서 연관성을 계산하는 방식. 예를들어 어린이의 나이와 키에 대한 데이터의 경우, 나이순위에 따라 키의 순위가 동일한 경우가 있음. |
| spearman 스피어먼 상관계수 |
두 변수의 순위 값 사이의 피어슨 상관 계수와 같음. 즉, 순서척도가 적용되는 경우에는 스피어먼 상관계수가, 간격척도가 적용되는 경우에는 피어슨 상관계수가 적용됨. |
# 2-1_데이터 왜도
- 왜도: 비대칭도 라고도 하며, 평균에 대해 최빈값이 얼마나 치우쳐져있는지를 나타내는 척도. 왜도는 우측으로 치우칠수록 음의값, 좌측으로 치우칠수록 양의 값을 가짐.
데이터가 얼마나 불균형한 분포를 나타내는지를 보여주는 수치인듯??
skew = data.skew()
print(skew)
# 2-2_데이터 분포 시각화
a) histogram
import matplotlib.pyplot as plt
data.hist(bins=20, grid=True, figsize=(12,8), color='#86bf91', rwidth=0.8)
plt.show()
데이터별 분포를 히스토그램으로 나타냄.
b) density plot
data.plot(kind='density', subplots=True, layout=(3,3), figsize = (15,15), sharex=False)
plt.show()
데이터셋에는 음수값은 없지만 시각적으로 더 나아보이는..?
c) box plot
먼저 box plot 형태의 기본적인 구조를 확인

| MAXIMUM 최대값 |
제 1사분위에서 1.5 IQR(데이터의 중간 값 (Q3 - Q1))을 뺀 값들 |
| UPPER QUARTILE 제 3사분위 수 (Q3) |
중앙값 기준으로 상위 50% 중의 중앙값, 전체 데이터 중 상위 25%에 해당하는 값 |
| MEDIAN 중위수 |
데이터의 정 가운데 순위에 해당하는 값들 |
| LOWER QUARTILE 제 1사분위 수 (Q1) |
중앙값 기준으로 하위 50% 중의 중앙값, 전체 데이터 중 하위 25%에 해당하는 값 |
| MINIMUM 최소값 |
제 3사분위에서 1.5 IQR을 더한 값들 |
| OUTLIER 이상치 |
수염 사이에 포함되지 않은 모든 값 |
d) confusion matrix
correlations = data.corr(method = 'pearson')
fig = plt.figure(figsize=(6,6))
ax = fig.add_subplot(111)
cax = ax.matshow(correlations, vmin=-1, vmax=1)
fig.colorbar(cax)
ticks = np.arange(0,9,1)
ax.set_xticks(ticks)
ax.set_yticks(ticks)
ax.set_xticklabels(names)
ax.set_yticklabels(names)
plt.show()
피어슨 상관 계수의 출력을 confusion matrix로 시각화
e) Scatter matrix
전체 데이터에 대한 Scatter plot을 matrix 형태로 출력
from pandas.plotting import scatter_matrix
scatter_matrix(data, figsize=(12, 12))
plt.show()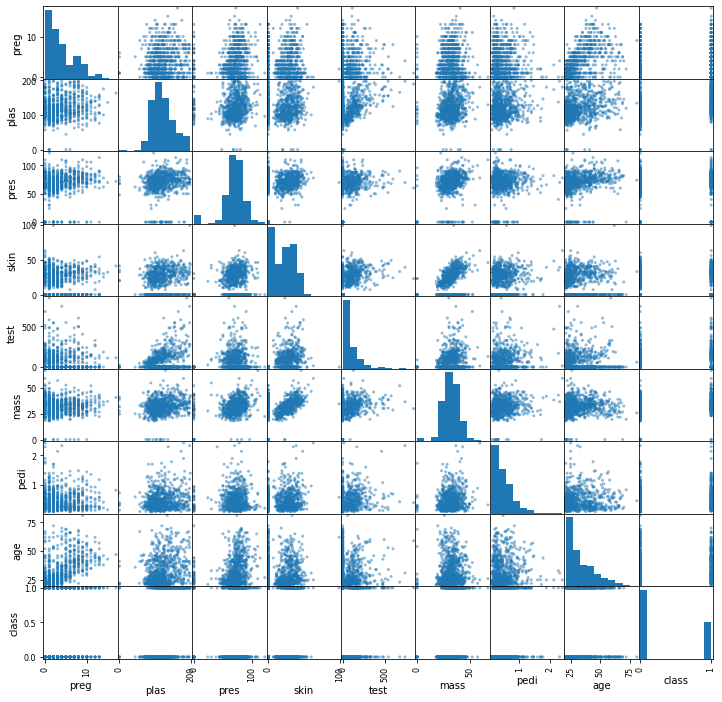
# 3_데이터 스케일링
이제 본격적으로 scikit learn을 활용하여 데이터 분석을 실시해본다.
a) MinMaxScaler
모든 값(음수 포함)을 원하는 값의 범위로 바꾸는 것.
import scipy
from sklearn.preprocessing import MinMaxScaler
array = data.values # pandas frame을 numpy array 형태로 변환
print(data.head())
print('---------------------------------------------------------------------------')
X = array[:,0:8] # Class 컬럼을 제외한 모든 값 리스트에 삽입
Y = array[:, -1] # Class 컬럼 값만 삽입(라벨)
print(X[0:5,:])
print('---------------------------------------------------------------------------')
scaler = MinMaxScaler(feature_range=(-1,1)) # 변환된 특징 값들의 범위를 -1 ~ 1 로 설정
rescaledX = scaler.fit_transform(X)
np.set_printoptions(precision=3)
print(rescaledX[0:5,:])
6 -> -0.294, 148 -> 0.487 등으로 값들이 정규화가 되었다.
b) StandardScaler
모든 값을 평균이 0, 분산이 1인 정규 분포로 바꾸는 것.
from sklearn.preprocessing import StandardScaler
X = array[:, 0:8]
Y = array[:, -1]
scaler = StandardScaler().fit(X)
rescaledX = scaler.transform(X)
np.set_printoptions(precision=3)
print(rescaledX[0:5, :])
c) Normalizer
모든 값(음수 포함)을 0~1 사이의 값으로 바꾸는 것.
from sklearn.preprocessing import Normalizer
X = array[:, 0:8]
Y = array[:, -1]
scaler = Normalizer().fit(X)
normalizedX = scaler.transform(X)
np.set_printoptions(precision=3)
print(normalizedX[0:5, :])
MinMaxScaler의 하위호환인듯?
d) Binarizer
모든 값을 특정 조건에 따라 0 또는 1의 값으로 바꾸는 것.
from sklearn.preprocessing import Binarizer
X = array[:, 0:8]
Y = array[:, -1]
binarizer = Binarizer(threshold=0.0).fit(X) # 0.0 초과시 1, 아니면 0
binaryX = binarizer.transform(X)
np.set_printoptions(precision=3)
print(data.head())
print('---------------------------------------------------------------------------')
print(binaryX[0:5, :])
여기까지 기본적인 상관 분석에 대한 것을 마치고 다음 챕터에서 머신러닝을 활용해보도록 한다.
'Python > Scikit learn' 카테고리의 다른 글
| [Scikit learn] 머신러닝 기초 잡기 - 2. 머신러닝 (0) | 2023.01.30 |
|---|

댓글